2021年8月9日,Nature Genetics在线发表了河北农业大学马峙英团队题为“High-quality genome assembly and resequencing of modern cotton cultivars provide resources for crop improvement”研究论文。该研究率先组装陆地棉现代品种农大棉8号和海岛棉Pima90高质量基因组,完成1,081份大规模陆地棉品种资源基因组深度重测序,破译海陆、陆陆基因组和国内外不同材料的结构变异,揭示了结构变异对纤维品质、产量、黄萎病抗性的遗传效应及其在基因型-表型关系中的重要作用,为作物多性状协同改良提供了新的理论依据和资源。

育种家在获得多逆境抗性的同时,一直努力通过协同提高产量和品质来培育陆地棉新品种,并将海岛棉的优异性状转育到陆地棉中以进一步改良品质和抗性。然而,人们对棉花现代育成品种的基因组信息、现代育种过程中海陆棉种间基因组变异仍缺乏深入了解。广泛的基因组结构变异(插入、缺失、倒位和易位)意味着任何一个单体型都可能缺失或者包含大多数群体中不存在的序列变异,基于更多基因组组装和品种资源重测序揭示的结构变异对于棉花分子改良非常重要,而有关棉花结构变异的规律和遗传效应还鲜为人知。
1. 首次组装了两个陆、海新品种基因组。该研究率先组装了我国自育陆地棉现代品种农大棉8号(NDM8)(优质高产多抗,2010-2017年6次进入全国大田作物授权品种面积前十排行榜)和海岛棉新品系Pima90(抗病优质,长期用于棉花抗病遗传、QTL定位和基因克隆及分子育种)的基因组。NDM8和Pima90的基因组重要质量参数:大小分别为2.29Gb和2.21Gb,Contig N50为13.15Mb和9.24Mb,染色体挂载率为99.57%和99.75%,gap比率为0.003%和0.06%,比对率均为99.16%,组装错误率为1.87×10-7和2.95×10-7,BUSCO评估结果为96.1%和95.9%,LAI值为14.2和12.1,表明组装了两个高质量基因组。鉴定出陆地棉NDM8蛋白编码基因80,124个,其中1,499个为预测的新基因;海岛棉Pima90基因79,613个,其中1,267个为预测的新基因。98%以上的基因都能被多样化转录组数据支持。分析还发现,在棉种进化中,Copia和Gypsy转座子对农艺性状的分化起着重要作用。
2. 破译了海岛棉、陆地棉结构变异图谱。为了在陆地棉现代育种中潜在有效地利用海岛棉基因组变异,将海岛棉Pima90基因组与陆地棉NDM8基因组比对分析,发现Pima90存在高度的基因组多样性,共检测到846,363个结构变异,包括517,230个插入、317,638个缺失、9,515个倒位和1,980个易位。插入和缺失在D亚组上的密度明显高于A亚组;31,296个变异-基因对(插入缺失位于基因内和/或调控区两侧±1kb)在海岛棉组织中显著特异表达;5,815个插入缺失位于5,256个基因的外显子区,其中3,178个变异与转录产物一致。发现Pima90中编码蔗糖合酶的基因GbM_D13G2394存在2bp的缺失,该缺失在海岛棉品种Hai7124和3-79基因组以及海岛棉渐渗系NDM373-9和鲁原343中也存在。GbM_D13G2394在海岛棉纤维伸长期和次生壁加厚期高表达,意味着该缺失对海岛棉纤维长度和强度有重要作用。追踪利用Pima90和CCRI8海陆回交育成的新材料NDM373-9的基因组序列,发现新品系获得了来自于海岛棉的171个外显子区结构变异,其中有34个和12个基因分别与已报道的抗病性和纤维发育有关。
为了探明陆地棉现代品种的基因组变化,将NDM8基因组和TM-1(Wang, M. J. et al., Nature Genetics, 2019)进行比较,发现NDM8存在876,568个结构变异,包括27,708个插入、47,221个缺失、808个倒位和831个易位,其中28,626个结构变异能够在10~1081个重测序种质材料中检测到。插入和缺失在D亚组上的密度明显高于A亚组,而且在端粒附近具有偏好性,是染色体其它区域的3.71倍,远高于Pima90(1.89倍)。发现603个插入缺失位于526个基因的外显子区,其中189个是TM-1的同源基因,76个是非同源基因,261个未注释到功能。同源基因中编码肉桂酰辅酶A还原酶的基因GhM_A02G1731在TM-1存在1个缺失,导致该基因的NAD结构域受损,影响了基因的抗病性功能。研究还发现,就倒位和易位长度而言,两个品种的育成年代相差越近,存在的倒位和易位变异越少。现代品种与早期品种相比,获得了1,128个NDM8基因组结构变异,而且D亚组较A亚组经历了更强的育种选择。
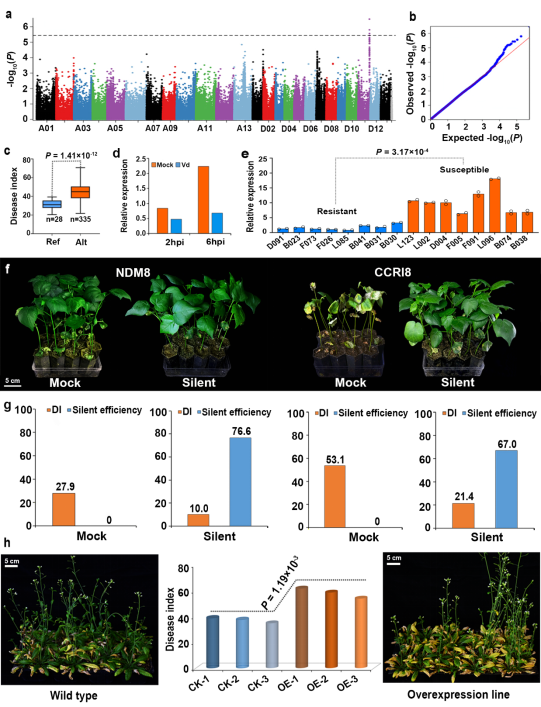
3. 揭示了棉花重要性状结构变异和基因。为了揭示基因组结构变异对重要性状的遗传效应,以现代品种NDM8为参考基因组,基于1,081份来自于世界各地的陆地棉种质资源重测序(平均10.65×)数据筛选获得的304,630个结构变异,以及大规模环境评价获得的纤维长度、比强度、马克隆值、铃重、衣分、子指等表型数据和401份资源的黄萎病抗性数据进行GWAS分析,发现346个与品质、97个与产量、3个与黄萎病抗性显著关联的重要结构变异。在Dt11染色体的1个370kb区域内(24.55~24.93Mb),有69个和56个NDM8型结构变异能够使纤维长度分别显著增加0.71~0.99mm和1.00~1.19mm,使得长度从27mm或者28mm级别增至29mm级别(纤维长度增加1mm,每吨皮棉售价一般可增加300元左右);在Dt03染色体上的2个变异能够分别使衣分从37.49%增至39.69%、37.47%增至40.00%;在Dt11染色体的69.00~69.33 Mb区段内的3个变异能够使黄萎病病情指数(DI)降低13.6,可使棉花抗性反应型从感病(DI=44.5~45.2)变为耐病(DI=30.9~31.1)。在同时被平均值和育种值关联到的193个变异中,与品质关联的变异主要位于D亚组,而产量性状的主要位于A亚组;在找到的907个与纤维品质、产量相关的基因中,84.23%的基因在纤维不同发育时期表达,基因内或调控区含有305个结构变异,预示这些基因潜在影响纤维品质和产量。在黄萎病抗性相关基因中,GhNCS编码S-去甲乌药碱合成酶,受大丽轮枝菌胁迫后该基因下调表达且在抗病品种中的表达量显著低于感病品种,在耐病品种NDM8和感病品种CCRI8中沉默该基因都导致抗病性显著增强,使NDM8由耐病变为抗病,CCRI8从感病变为耐病,然而拟南芥中过量表达该基因表现为更感病,表明GhNCS是控制黄萎病抗性的一个重要新基因。
该研究得到国家重点研发计划(2016YFD0101405和2016YFD0101006)、国家棉花产业技术体系(CARS15-03)、河北省科技支撑计划(16226307D)和河北省高端人才计划(031601801)资助。
原文链接:https://www.nature.com/articles/s41588-018-0119-7






